概要
ヒト由来試料(皮膚、糞便など)、環境水、土壌、食品などの幅広い試料に存在する微生物叢(Microbiome、マイクロバイオーム)の遺伝子解析を行い、系統分類、同定を行うサービスです。(
fig.1)
特長
- 微生物叢標準品および、試料の前処理・保存、核酸精製、NGSライブラリーの構築の各ステップにZymoBIOMICSシリーズの高品質かつ検証済みの微生物叢研究用試薬を用いることで、バイアスや外来性DNAの混入がなく、信頼性の高い試料調製を行えます。
- 調製した試料のNGS(次世代シークエンシング)解読後、最先端のバイオインフォマティクスで解析します。
- 論文に使用可能なデータ形式で、解析結果を納品いたします。
- レポートは、プロジェクトまたはアプリケーションに応じて完全にカスタマイズが可能です。
サービス一覧
| サービス名 |
対象生物 |
シークエンシングのプラットフォーム |
16Sシークエンシングサービス
(V1-V2、V3-V4、V4、or V6-V8) |
細菌、古細菌 |
Illumina MiSeq、NextSeq 2000 |
| ITSシークエンシングサービス |
真菌類 |
Illumina MiSeq、NextSeq 2000 |
| 18Sシークエンシングサービス |
真核生物 |
Illumina MiSeq、NextSeq 2000 |
| ショットガンメタゲノムサービス |
細菌、ウイルス、真菌類、原生生物 |
Illumina社NovaSeq 6000、NestSeq 2000、NestSeq X |
16Sシークエンシングサービス
細菌、古細菌について種レベルでの同定が可能です。
ZymoResearch社 16Sシークエンシングサービスおよび既存法(QIIMEソフトウェアとGreengenesデータベースの組み合わせ)による種レベルでの同定可否(
fig.2)
ショットガンメタゲノムサービス(fig.3)
- CosmosID社との提携により、業界最先端の感度と精度で解析を行います。
- すべての微生物界(細菌、古細菌、ウイルス、原生生物、真菌)について、株(strain)レベルでの同定を行えます。
- 用いているデータベースは、15万以上のゲノムと遺伝子配列、1億のバイオマーカーを有しています。
- データ構造はゲノムの系統学的階層に従っており、未知の種についても、仮想の系統樹上で正確に分類することができます。
納品されるレポートの例
下記のレポートはすべて、10種類のヒト糞便試料(S1~S10)を16Sシークエンシングで解析したデータです。
| 解析項目 |
概要 |
レポート例 |
| 微生物組成プロット(棒グラフ) |
|
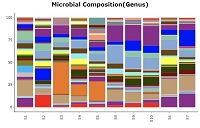
|
| 微生物の分類群ごとの存在度ヒートマップ |
- Bray-Curtisの種組成類似度指数を用いたクラスター解析による微生物の分類群毎の存在度ヒートマップは、試料間での微生物分布のパターンを簡単に特定できる。
|

|
| 固有配列の存在度ヒートマップ |
- シークエンシングのrawデータから推測される固有配列の存在度ヒートマップ。DADA2プログラムを用いることにより、2つの試料間での1塩基の違いも識別することができる。
- 例えば、16S V3-V4領域の2bpが異なる2つの固有配列について、同じ分類に割りあてられている可能性がある。この場合、2つの亜種の存在を示している可能性がある。そのため、分類群的な割り当てを超えて、固有配列の分布を解析する必要がある。
|

|
| α多様性の解析 |
- α多様性は、各試料における微生物多様性の尺度。
- 通常、シークエンス深度がより深くなると、より存在度が少ない微生物が識別されるようになり、α多様性が増加する。α多様性希薄化曲線は、この傾向を説明するのに適した方法。
|

|
| β多様性の解析 |
- β多様性は、試料間における微生物多様性の違いの尺度。
- Bray-Curtisの種組成類似度指数により求められた試料間のペアワイズ距離をプロットしている。
- 図の各点は、全体の微生物組成プロファイル。類似の微生物組成プロファイルを有する試料は互いに近く位置する。
|
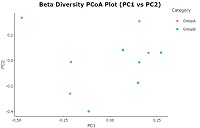
|
| LEfSe解析 |
- LEfSe(Linear discriminant analysis effect size)解析は、微生物分布が定義済みのグループ間で、有意かつ統計的に微生物分布が異なる場合の分類群の識別に有用。
|
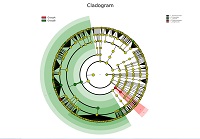
|
| Taxa2SV Decomposerを用いた固有配列の解析 |
- 「興味がある分類群が固有配列を有するか。」「その固有配列と、参照データベースの代表配列がどのように比較されるか。」などの解析に有用。
- データは分類群(門、属、種)毎に分けて出力されており、興味がある分類群について解析を行える。
|
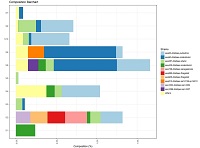
|
使用文献
"Analysis of Fusobacterium persistence and antibiotic response in colorectal cancer.",
Bullman, S., et al., Science, 358 (6369), 1443~1448 (2017) [PMID: 29170280]